Библиотека для реализации генетических алгоритмов в .NET Framework 2.0
Содержание
- Файлы
- Теория
- Реализация
- Общий план использования библиотеки
- Базовый класс особей для работы с дробными хромосомами
- Общий план использования библиотеки с применением класса BaseDoubleSpecies
- Класс Analytics
- Примеры использования
- История версий
- Комментарии
Файлы
Теория
Для начала надо сказать, что из себя представляют эти самые генетические алгоритмы. Подробно описывать их не буду, т.к. вы можете найти много хороших статей по ним и интернете. А если в двух словах, то генетический алгоритм - это такой алгоритм с помощью которого можно искать минимум (или максимум) целевой функции, зависящей от многих переменных. Такой алгоритм применяют, когда целевая функция сложная и не понятно как еще можно найти ее экстремум. Для примера, целевой функцией может быть ошибка между полученными данными и расчетными с параметрами, которые надо установить. Например, в исходниках, которые вы можете скачать по ссылке в конце статьи, алгоритм вычисляет коэффициенты перед переменными.
Идея генетических алгоритмов взята из теории Дарвина о эволюции. Мы создаем виды (решения), которые скрещиваются между собой, мутируют, самые неудачные умирают. Суть алгоритма в том, что изначально мы имеем набор произвольных видов. Каждый вид содержит в себе набор хромосом (переменных, значение которых надо найти), которые и надо рассчитать. Сначала мы имеем в популяции виды, у которых все хромосомы случайны. После этого происходит скрещивание видов и, возможно, мутирование. После этого отбираются самые лучшие виды (у которых минимальна целевая функция), а самые плохие (с максимальными целевыми функциями и с хромосомами, которые не попадают в заданный интервал) удаляются из популяции. На следующей итерации скрещивание, мутирование и отбор повторяется. Благодаря этому у нас постепенно остаются только те, у которых целевая функция близка к минимуму. И так повторяется до тех пор пока не будет найдено решение, удовлетворяющее нас с точки зрения погрешности. Также часто алгоритм останавливают, если в течение заданного числа поколений (итераций) не удается найти еще лучший вид, чем тот, что у нас есть.
Реализация
Реализация алгоритма была сделана на языке C# для .NET Framework 2.0.
Базовый класс для видов
Все виды должны быть производными от базового класса BaseSpecies<TSpecies>, где TSpecies - конкретный тип для вида. TSpecies должен быть производным от класса BaseSpecies<TSpecies>.
/// Базовый класс для вида
/// </summary>
abstract public class BaseSpecies<TSpecies>: IComparable
where TSpecies: BaseSpecies<TSpecies>
{
#region Статические функции для скрещивания хромосом базовых типов
/// <summary>
/// Скрестить две хромосомы типа double
/// </summary>
/// <param name="x">1-я хромосома</param>
/// <param name="y">2-я хромосома</param>
/// <returns>Новая хромосома</returns>
static protected double Cross (double x, double y)
{
...
}
static protected Int32 Cross (Int32 x, Int32 y)
{
...
}
static protected Int64 Cross (Int64 x, Int64 y)
{
...
}
#endregion
#region Методы для мутаций в базовых типах
/// <summary>
/// Мутация для типа double
/// </summary>
/// <param name="val">Мутируемое значение</param>
/// <returns>Промутирующее значение</returns>
static protected double Mutation (double val)
{
...
}
static protected Int32 Mutation (Int32 val)
{
...
}
static protected Int64 Mutation (Int64 val)
{
...
}
#endregion
/// <summary>
/// Мертв ли вид.
/// </summary>
/// <remarks>Например, когда хромосовы не попадают в заданные интервал</remarks>
protected Boolean m_Dead = false;
/// <summary>
/// Мертвый ли вид?
/// </summary>
public bool Dead
{
get { return m_Dead; }
}
/// <summary>
/// Проверяет, чтобы все хромосомы попали бы в заданные интервалы.
/// В противном случае помечает вид как "мертвый".
/// </summary>
abstract public void TestChromosomes();
/// <summary>
/// Метод для скрещивания себя с другим видом
/// </summary>
/// <param name="Species">Другой вид</param>
/// <returns>Полученный вид</returns>
abstract public TSpecies Cross (TSpecies Species);
/// <summary>
/// Произвести мутацию
/// </summary>
abstract public void Mutation();
/// <summary>
/// Целевая функция. Должна в случае удачного набора хромосом стремиться к своему минимуму
/// </summary>
/// <returns>Значение целевой функции</returns>
abstract public double FinalFunc
{
get;
}
#region IComparable Members
/// <summary>
/// Вид считается больше, если он мертв или у него больше целевая функция
/// </summary>
/// <param name="obj"></param>
/// <returns></returns>
public Int32 CompareTo(object obj)
{
...
}
#endregion
}
Как видно из кода, BaseSpecies - это абстрактный класс, который содержит статические protected-методы для скрещивания и мутации. Разберем некоторые методы поподробнее. Начнем с методов, которые необходимо реализовать в производных классах.
Этот методы должен устанавливать внутреннюю защищенную Булеву переменную m_Dead, которая показывает, подходят ли хромосомы по ограничениям, которые на них накладывают (например, в нашем примере, попадают ли значения хромосом в отрезок [-5.0; 5.0]). Если значения хромосом нас устраивают, то m_Dead надо установить в false, иначе в true. По этой переменной (получаемую через свойство Dead) популяция отбрасывает заведомо неудачные виды.
Следующий метод, который необходимо реализовать в производном классе, это
Этот метод создает новый вид, скрещивая себя и другой вид, который передается как аргумент. Здесь надо сделать некоторые пояснения. Обычно, если вид содержит несколько хромосом, скрещивают хромосомы "одного вида", т.е. в нашем случае скрещиваем a0 себя (т.е. this) и a0 другого вида, аналогично скрещиваем a1 и a1, т.е. нет смысла скрещивать a0 себя и a1 другого класса. Возможно, у вас будет задача, где это понадобится, но я так сходу ее придумать не смогу.
Смысл скрещивания состоит в том, что берем одну часть хромосомы себя и вторую часть другого вида и создаем из них новую хромосому, которая и будет содержаться в новом виде. Часто хромосомы скрещивают побитово. Суть его состоит в том, что сначала случайно выбираем точку разрыва (скрещивания) хромосомы, потом создаем новую хромосому, которая состоит из левой части первой хромосомы и правой второй. Пусть например у нас есть две хромосомы (8-битовые для простоты): 10110111 и 01110010. Случайно выбираем точку разрыва (отмечена символом '|'):
101 | 10111
011 | 10010
Отсюда мы может сделать две разные хромосомы это 101 10010 и 011 10111. Какую из них выбрать - это также можно решить генератором случайных чисел. Также можно искать две и более точек пересечения. Таким образом, мы должны иметь конструктор, который создает вид из хромосом.
Для того, чтобы не надо было изобретать велосипед, в классе BaseSpecies есть публичные статические методы для скрещивания хромосом некоторых типов (Double, Int64 и Int32). Разберем поподробнее первые два метода. В данном случае по сути есть две точки пересечения - в середине слова и знак числа, т.е. сначала скрещиваются хромосомы побитово без учета знака, а потом также случайно берем знак от одной из хромосомы.
Рассмотрим код.
/// Скрестить две хромосомы типа double
/// </summary>
/// <param name="x">1-я хромосома</param>
/// <param name="y">2-я хромосома</param>
/// <returns>Новая хромосома</returns>
static protected double Cross (double x, double y)
{
Int64 ix = BitConverter.DoubleToInt64Bits(x);
Int64 iy = BitConverter.DoubleToInt64Bits(y);
double res = BitConverter.Int64BitsToDouble(BitCross(ix, iy));
if (m_Rnd.Next() % 2 == 0)
{
if (x * res < 0)
{
res = -res;
}
}
else
{
if (y * res < 0)
{
res = -res;
}
}
return res;
}
Работает этот метод следующим образом: сначала преобразуем хромосомы из типа Double в Int64. Это сделано для того, чтобы можно было бы без проблем скрещивать побитово. (Спасибо henson-у с форума RSDN за то, что подсказал, что есть метод DoubleToInt64Bits(). Собственно, все эта ветка форума находится здесь.) После скрещивания уже типов Int64 без учета знака (об этом чуть позже), переводим число обратно к Double, а потом берем знак одной из двух хромосом. Также я видел реализацию генетического алгоритма, где для скрещивания дробных хромосом просто брали их среднее арифметическое, однако в тех, примерах, которые я пробовал, такой метод работает лучше.
А теперь давайте посмотрим как происходит скрещивание типов Int64:
/// Скрестить побитово без учета знака две хромосомы типа Int64
/// </summary>
/// <param name="x">1-я хромосома</param>
/// <param name="y">2-я хромосома</param>
/// <returns>Новая хромосома</returns>
static protected Int64 BitCross (Int64 x, Int64 y)
{
// Число бит, оставшиеся слева от точки пересечения хромосом
int Count = m_Rnd.Next(62) + 1;
Int64 mask = ~0;
mask = mask << (64 - Count);
return (x & mask) | (y & ~mask);
}
/// <summary>
/// Скрестить побитово с учетом знака две хромосомы типа Int64
/// </summary>
/// <param name="x">1-я хромосома</param>
/// <param name="y">2-я хромосома</param>
/// <returns>Новая хромосома</returns>
static protected Int64 Cross (Int64 x, Int64 y)
{
Int64 res = BitCross(x, y);
if (m_Rnd.Next() % 2 == 0)
{
if (x * res < 0)
{
res = -res;
}
}
else
{
if (y * res < 0)
{
res = -res;
}
}
return res;
}
Как видно из кода, сначала скрещиваются хромосомы без учета знака, а потом (как и с типом Double) выбирается знак одной из хромосом. Точнее, это работает так, что сравнивают знак выбранной хромосомы с результатом, и, если знаки не совпали (произведение меньше нуля), то знак результата меняется на противоположный. Скрещивание без учета знаков - это просто игра с битами.
Если вас по каким-то причинам не устраивают эти методы для скрещивания, то вы можете сделать свои и использовать их, а мы переходим к следующему абстрактному методу, который надо реализовать.
public void Mutation(); Здесь все очень похоже на скрещивание. Мутация действует на одну хромосому. В теории при мутации могут происходить любые изменения. Но в данной реализации мутация меняет один бит в слове. Вот пример кода:
/// Мутация для типа double
/// </summary>
/// <param name="val">Мутируемое значение</param>
/// <returns>Промутирующее значение</returns>
static protected double Mutation (double val)
{
UInt64 x = BitConverter.ToUInt64(BitConverter.GetBytes(val), 0);
UInt64 mask = 1;
mask <<= m_Rnd.Next(63);
x ^= mask;
double res = BitConverter.ToDouble(BitConverter.GetBytes(x), 0);
return res;
}
/// <summary>
/// Мутация для типа Int64
/// </summary>
/// <param name="val">Мутируемое значение</param>
/// <returns>Промутирующее значение</returns>
static protected Int64 Mutation (Int64 val)
{
Int64 mask = 1;
mask <<= m_Rnd.Next(63);
return val ^ mask;
}
Мутации происходят сравнительно редко. Например, часто вероятность мутации делают 5-10%, но иногда стоит попробовать сделать ее побольше (примерно 30%). Также довольно часто в качестве мутации для дробных чисел лучше применять не написанную выше функцию, а просто генератор случайных чисел, который выдает случайное число в нужном нам интервале. В прилагаемом примере для дробных чисел так и сделано, а для целых хромосом используется мутация, описанная здесь.
Реализация популяции (класс Population)
А теперь рассмотрим класс популяции, где живут, размножаются и умирают виды. Вы будете добавлять свои виды в этот класс (точнее в массив видов, находящийся в этом классе). Класс Population объявлен как
Как видно, он имеет generic-параметр, который представляет собой вид, который должен быть производным от BaseSpecies<TSpecies>
Начнем со свойств, которые надо настроить перед началом работы алгоритма.
- MaxSize (Int32) - максимальное число видов, которое может содержать популяция. Это тот размер, до которого "урезается" популяция после скрещивания. По умолчанию 500.
- CrossPossibility (Double) - вероятность скрещивания. Она должна быть в интервале (0.0; 1.0]. Если это условие не выполняется, то бросается исключение ArgumentOutOfRangeException. По умолчанию это значение установлено в 0.95.
- MutationPossibility (Double) - вероятность мутации. Она должна быть в интервале [0.0; 1.0). Если это условие не выполняется, то бросается исключение ArgumentOutOfRangeException.
В принципе, можно было бы ввести еще вероятность отбора, но в данном случае она считается равной 1.0. В противном случае надо было бы удалять виды из популяции не все подряд самые плохие, а с некоторой вероятностью. Но мертвые виды (напомню, что это виды, у которых хромосомы не попадают в заданный интервал или не удовлетворяет другим условиям) надо удалять в любом случае.
А теперь рассмотрим публичные методы, которые необходимо вызывать.
public void Add (TSpecies species) - добавить новый вид в популяцию. Заметьте, что вы должны вручную добавлять необходимое число видов. Перед началом работы алгоритма надо, чтобы в популяции было хотя бы два вида. Число видов в популяции может быть меньше, чем установленное значение MaxSize. Если после скрещивания размер популяции меньше MaxSize, то просто не удаляются самые плохие виды (даже мертвые).
Чтобы получить следующую популяцию, необходимо вызвать метод void NextGeneration(). Работа этого метода может занимать достаточно много времени, поэтому лучше всего его вызывать из отдельного потока. Давайте посмотрим что он делает.
/// Обновить популяцию (получить слудующее поколение)
/// </summary>
public void NextGeneration()
{
if (m_Generation == 0 && m_Species.Count == 0)
{
throw new ZeroSizePopulationException();
}
// Сначала скрестим
Cross();
// Промутируем и заодно проверим все хромосомы
foreach (BaseSpecies<TSpecies> species in m_Species)
{
// Если надо мутировать с учетом вероятности.
if (m_Rnd.NextDouble() <= m_MutationPossibility)
{
species.Mutation();
}
species.TestChromosomes();
}
// Отберем самые живучие виды
m_Species.Sort();
Selection();
m_Generation++;
}
Сначала метод проверяет, чтобы при первом вызове метода (при нулевом поколении, о поколениях чуть позже) размер популяции был бы больше 2 (иначе некого скрещивать). Если это не так, то бросается исключение SmallSizePopulationException. После этого происходит скрещивание видов:
/// Получить новые виды скрещиванием
/// </summary>
protected void Cross()
{
// Размер до начала пополнения популяции (чтобы не скрещивать новые виды,
// которые добавляются в конец списка)
Int32 OldSize = m_Species.Count;
// Номер пары для скрещиваемого вида
Int32 Count = m_Species.Count;
for (int i = 0; i < Count; ++i)
{
// Если надо скрещивать с учетом вероятности.
if (m_Rnd.NextDouble() <= m_CrossPossibility)
{
// Добавляем в список вид, полученный скрещиванием очередного вида и
// вида со случайным номером.
m_Species.Add (m_Species[i].Cross (m_Species[m_Rnd.Next (OldSize)] ) );
}
}
}
При скрещивании перебираем все виды и скрещиваем их с установленной вероятностью скрещивания с другими видами, которые выбираются случайно.
После скрещивания происходит мутация видов также с заданной вероятностью, а после скрещивания идет тест хромосом. Затем сортируем виды по значению их целевой функции. Метод Sort определен в классе BaseSpecies, здесь я его приводить не буду, но скажу, что вид считается больше тот, который мертвый, а, если все виды живые, то с максимальной целевой функцией. Отбор видов также происходит просто:
/// Произвести отбор самых "живучих" видов
/// </summary>
protected void Selection()
{
// Сколько видов надо удалить
Int32 Count = m_Species.Count - m_MaxSize;
for (Int32 i = 0; i < Count; ++i)
{
m_Species.RemoveAt (m_Species.Count - 1);
}
}
Общий план использования библиотеки
Рассмотрим общий план того, что необходимо сделать, чтобы использовать эту библиотеку:
- Создать класс вида, производный от BaseSpecies<TSpecies>, где в качестве аргумента обобщения в BaseSpecies передается имя самого класса вида.
- Внутри класса вида создать набор хромосом необходимых типов.
- Перегрузить абстрактный метод public TSpecies Cross (TSpecies Species), который должен создать новый вид. В качестве параметра метода выступает особь того же вида, с которым необходимо провести скрещивание.
- Перегрузить свойство (только get) public double FinalFunc(), которое возвращает значение целевой функции. Если целевая функция сложная, то удобно хранить значение целевой функции в отдельной переменной, значение которой и возвращает это свойство, а сам расчет целевой функции осуществлять в конструкторе вида.
- Перегрузить метод public void Mutation().
- Перегрузить метод public override void TestChromosomes(), который устанавливает значение переменной m_Dead в true, если данный вид нас заведомо не устраивает, например, е значения его хромосом не попадает в заданные пределы, и в false в противном случае.
- Создать популяцию Population<TSpecies>, которая будет хранить особей нужного вида.
- Вызвать метод Reset() популяции для удаления всех видов, если они уже были в популяции.
- Через свойство MaxSize популяции установить максимальный размер популяции.
- Установить вероятность мутации через свойство MutationPossibility. Значение этого свойство должно лежать в пределах от 0 до 1. Значение по умолчанию 0.1.
- Установить вероятность скрещивание через свойство CrossPossibility. Значение этого свойство должно лежать в пределах от 0 до 1, причем нулевая вероятность скрещивания не допускается. Значение по умолчанию 0.95.
- Добавить в популяцию необходимое число особей соответствующего вида через метод void Add (TSpecies).
- Вызывать метод void NextGeneration() популяции пока в этом будет необходимость, например, пока не будет достигнута нужная погрешность.
- Через свойство TSpecies BestSpecies можно получить лучшую на данной итерации (в данном поколении) особь вида, у которой целевая функция меньше, чем у остальных.
Базовый класс особей для работы с дробными хромосомами
Базовый класс BaseSpecies разрабатывался таким образом, чтобы как можно больше абстрагироваться от конкретной реализации особей, от того что из себя представляют хромосомы. Это могут быть дробные и целые числа, массивы или экземпляры других классов.
Однако, часто генетические алгоритмы используются, когда нужно минимизировать какую-нибудь математическую функцию, зависящую от некоторого количества переменных дробного типа. В этом случае хромосомы удобно представлять в виде массива дробных чисел, где каждый элемент массива - это одна хромосома. При этом будем считать, что размер этого массива остается постоянным для всех особей на протяжении всего времени работы алгоритма.
Именно для этой цели и был создан класс BaseDoubleSpecies. Благодаря нему можно существенно сократить код, который должен написать программист, реализующий алгоритм. Рассмотрим этот класс.
Объявление его довольно запутанное:
BaseSpecies<BaseDoubleSpecies<TSpecies>>, ICloneable
where TSpecies : BaseDoubleSpecies<TSpecies>
Здесь, как и раньше, TSpecies - это конкретный тип особей.
Класс BaseDoubleSpecies является абстрактным, но он реализует все, что должен реализовать класс особей, однако добавляет свой абстрактный метод, внутри которого и должен происходить расчет целевой функции. Эта функция называется CalcFinalFunc(), она должна возвращать тип double.
Чтобы при каждом сравнении особи с другой особью не приходилось вычислять значение целевой функции, она вычисляется один раз в конструкторе особи. Рассчитанное значение целевой функции сохраняется в переменной m_FuncVal.
Также класс BaseDoubleSpecies содержит следующие массивы:
- m_Chromosomes - хромосомы. Для доступа к этому члену предусмотрено свойство Cromosomes.
- m_Intervals - статический массив классов Interval. С помощью него задаются интервалы изменения каждой хромосомы. Для доступа к этому массиву предусмотрено свойство Intervals. Это свойство нужно устанавливать до того как начнет заполняться популяция. Если при создании особи какая-то хромосома не попадает в свой интервал, то особь помечается как мертвая. Размер этого массива всегда должен равняться размеру массива m_Chromosomes. Более того, при создании особи со случайным значением хромосом, именно из размера этого массива вычисляется количество хромосом.
Класс BaseDoubleSpecies реализует все этапы генетического алгоритма, которые не были реализованы в базовом классе BaseSpecies такие как скрещивание и мутацию. Класс BaseDoubleSpecies имеет два конструктора:
public BaseDoubleSpecies (double[] chromosomes)
Первый из них создает особь со случайными значениями хромосом, что удобно использовать при начальном заполнении популяции. Второй конструктор используется внутри самого класса BaseDoubleSpecies для операций скрещивания и мутации.
Общий план использования библиотеки с применением класса BaseDoubleSpecies
- Создать класс вида, производный от BaseDoubleSpecies<TSpecies>, где в качестве аргумента обобщения классу BaseDoubleSpecies передается имя самого класса вида.
- Перегрузить абстрактный метод CalcFinalFunc(), который должен возвращать значение целевой функции. Хромосомы при этом берутся из массива m_Chromosomes.
- Через статическое свойство Intervals класса особей установить интервал изменения хромосом.
- Создать популяцию Population<BaseDoubleSpecies<TSpecies>>, которая будет хранить особей нужного вида;
- Вызвать метод Reset() популяции для удаления всех видов, если они уже были в популяции;
- Через свойство MaxSize популяции установить максимальный размер популяции;
- Установить вероятность мутации через свойство MutationPossibility. Значение этого свойство должно лежать в пределах от 0 до 1. Значение по умолчанию 0.1;
- Установить вероятность скрещивание через свойство CrossPossibility. Значение этого свойство должно лежать в пределах от 0 до 1, причем нулевая вероятность скрещивания не допускается. Значение по умолчанию 0.95;
- Добавить в популяцию необходимое число особей соответствующего вида через метод void Add (TSpecies)
- Вызывать метод void NextGeneration() популяции пока в этом будет необходимость, например, пока не будет достигнута нужная погрешность;
- Через свойство TSpecies BestSpecies можно получить лучшую на данной итерации (в данном поколении) особь вида, у которой целевая функция меньше, чем у остальных.
Класс Analytics
В версии 2.2 появился новый класс Analytics, который может быть полезен для наблюдения за работой генетического алгоритма. Этот класс предназначен для сохранения лучшей особи из каждого поколения. Класс Analytics предназначен только для работы с классами, производными от BaseDoubleSpecies.
Пользоваться классом Analytics очень просто, для этого нужно выполнить следующие действия:
- Создать экземпляр класса Analytics<TSpecies>, где TSpecies - это класс, производный от BaseDoubleSpecies.
- Перед запуском алгоритма очистить статистику с помощью метода Clear().
- Затем на каждой итерации алгоритма (на каждом поколении) вызывать метод Add(), передавая ему экземпляр лучшей особи на данный момент.
- После этого в процессе расчета можно обращаться к списку сохраненных особей через свойство BestSpecies.
- Кроме того, используя метод ToString(), можно сохранить историю в виде текстовой таблицы, где каждая строка будет соответствовать одному поколению, а каждый столбец - одной дробной хромосоме. Последний столбец соответствует значению целевой функции.
Пример использования этого класса вы можете найти в проекте SchwefelTest, о котором рассказывается ниже.
Примеры использования
Рассмотрим задачу, которая решается в примерах, которые прилагаются в исходниках.
Проект RealTest
Начнем с проекта RealTest, где в качестве базового класса особей используется класс BaseSpecies.
Есть функция f = a5 * X05 + a4 * X14 + a3 * X23 + a2 * X32 + a1 * X4 + a0. Здесь X0...X4 - переменные функции, а a0...a5 - это коэффициенты, которые в принципе не меняются, но нам не известны и которые мы хотим найти. В проекте находятся два примера. Первый, когда значения коэффициентов a0...a5 являются дробными, а второй, когда они являются целыми значениями. Т.к. по сути эти примеры мало отличаются, то далее более подробно я буду рассматривать именно виды с дробными значениями хромосом.
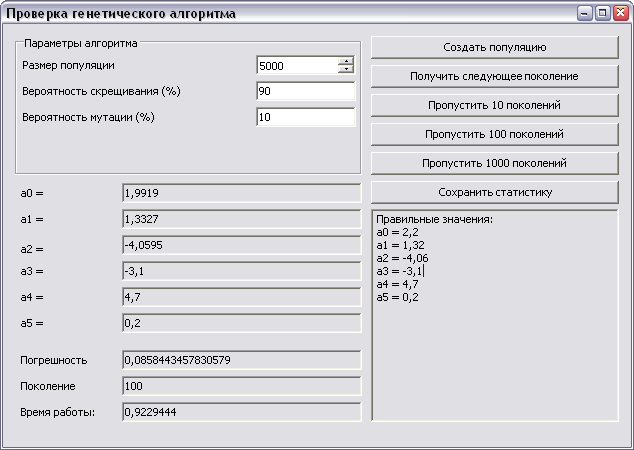
Изначально a0 = 2.2, a1 = 1.32, a2 = -4.06, a3 = -3.1, a4 = 4.7, a5 = 0.2. Для расчетов мы знаем значения функции в некоторых точках (30 точек). Для увеличения точности и скорости ограничим интервал, в котором находятся коэффициенты, отрезком [-5.0; 5.0].
Реализация видов
Я не буду приводить здесь полностью код, т.к. он довольно большой, а только его части. Вкратце опишу его работу, т.к. базовый класс оставляет довольно большую свободу выбора способа представления хромосом и данных.
Для начала объявлен класс для представления точек функции:
{
public const int FuncSize = 5;
/// <summary>
/// X1, X2, ..., X5
/// </summary>
private double[] m_X = new double[FuncSize];
public double[] X
{
get
{
return m_X;
}
}
/// <summary>
/// Значение функции
/// </summary>
private double m_Y;
public double Y
{
get
{
return m_Y;
}
}
public DoubleFuncPoint(double[] x, double y)
{
m_X = (double[])x.Clone();
m_Y = y;
}
}
Здесь я думаю все понятно. Затем создадим класс вида:
{...}
В самом классе вида DoubleSpecies (причем как статический член, чтобы у всех видов были одинаковые данные и не надо было бы их лишний раз передавать) хранится List<DoubleFuncPoint>, в который заносятся экземпляры класса DoubleFuncPoint. Значение целевой функции (что она из себя представляет расскажу чуть позже) вычисляется только в двух случаях для каждого вида, когда он создается и когда мутирует, а потом сохраняется в приватном поле и извлекается оттуда, когда это необходимо.
А целевая функция представляет из себя сумму квадратов разности значения функции, у которой известен ее вид и значением такой же функции, где вместо коэффициентов подставлены хромосомы вида:
{
m_FuncVal = 0.0;
foreach (DoubleFuncPoint point in m_FuncPoints)
{
double f = Func(point.X) - point.Y;
m_FuncVal += f * f;
}
}
Также еще без комментариев приведу методы для скрещивания и теста хромосом:
{
if (this == Species)
{
return new DoubleSpecies ((double[])m_Chromosomes.Clone());
}
DoubleSpecies Other = (DoubleSpecies)Species;
//В данном конкретном случае лучше работает скрещивание сразу всех хромосом
double[] chromosomes = new double[m_Chromosomes.Length];
for (int i = 0; i < chromosomes.Length; ++i)
{
chromosomes[i] = Cross(m_Chromosomes[i], Other.Cromosomes[i]);
}
return new DoubleSpecies(chromosomes);
}
public override void TestChromosomes()
{
Boolean res = false;
foreach (double chromosome in m_Chromosomes)
{
if (Double.IsNaN(chromosome) || chromosome < m_MinVal || chromosome > m_MaxVal)
{
res = true;
break;
}
}
m_Dead = res;
}
При скрещивании в нашем случае есть два варианта: скрещивать сразу все хромосомы или скрещивать по одной хромосоме за один раз. В данном примере лучше (быстрее находится минимум), если скрещивать сразу все хромосомы, а в другом случае, например, как в примере с целыми хромосомами лучше работает скрещивание по одной хромосоме. Здесь уже надо экспериментировать.
Работа примера
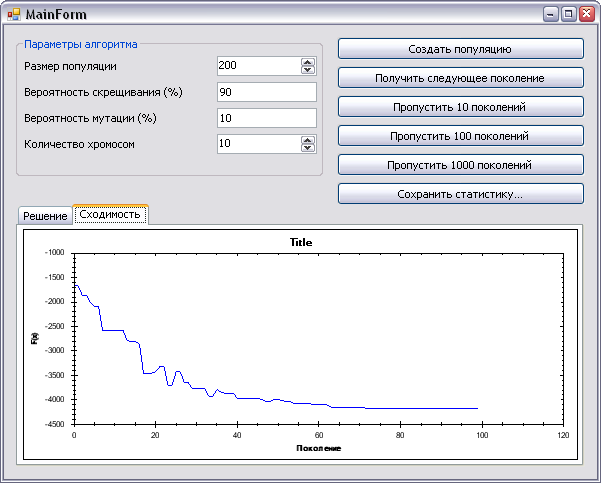
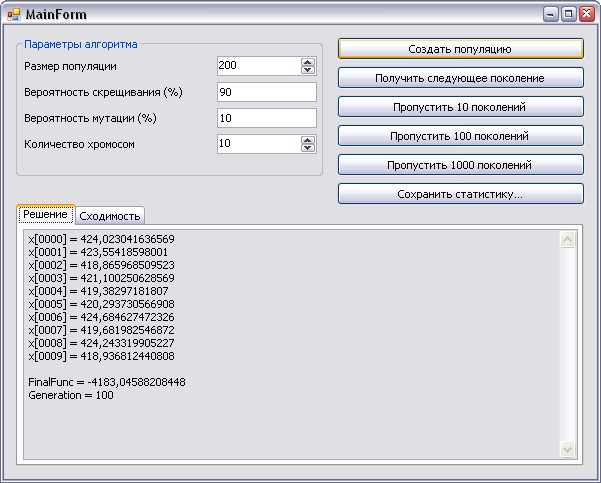
После компиляции примера, его запуска и выбора типа хромосом (дробные или целые) откроется окно, в котором можно установить параметры для генетического алгоритма и смотреть как изменяются значения хромосом лучшего вида, а также ошибка. Для простоты реализации весь расчет происходит в одном потоке. Пример работы генетического алгоритма с конкретными параметрами виден на рисунке. Напомню правильные значения: a0 = 2.2, a1 = 1.32, a2 = -4.06, a3 = -3.1, a4 = 4.7, a5 = 0.2.
Проект SchwefelTest
Этот проект реализует минимизацию функции Швефеля, формула которой представлена ниже
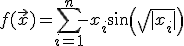
Эта функция замечательна тем, что она имеет много локальных минимумов и максимумов, но глобальный минимум у нее только один. Если все n переменных 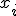 изменяются в интервале (-500.0; 500.0), то глобальный минимум будет в точке, когда все переменные
изменяются в интервале (-500.0; 500.0), то глобальный минимум будет в точке, когда все переменные 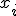 будут равны 420.9687. При этом значение целевой функции будет равно -n · 418.9829.
будут равны 420.9687. При этом значение целевой функции будет равно -n · 418.9829.
Чтобы иметь представление о том насколько изрезана целевая функция, на следующем рисунке приведены линии уровня для n = 2.
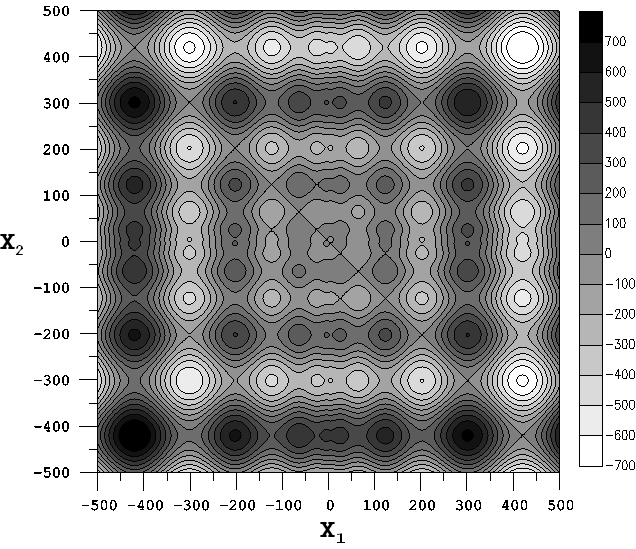
Искомый минимум находится в правом верхнем углу, но при желании можно искать и максимум, который находится в левом нижнем углу.
Этот пример показывает насколько сокращается код особей, если использовать класс BaseDoubleSpecies. Я не буду приводить полностью код проекта, приведу только код особи и код подготовки алгоритма:
{
public SchwefelSpecies ():
base ()
{
}
public SchwefelSpecies (double[] chromosomes):
base (chromosomes)
{
}
protected override double CalcFinalFunc ()
{
double result = 0;
for (int i = 0; i < m_Chromosomes.Length; i++)
{
result -=
m_Chromosomes[i] * Math.Sin (Math.Sqrt (Math.Abs (m_Chromosomes[i]) ) );
}
return result;
}
}
Согласитесь, что так особь выглядит намного приятнее.
А вот как выглядит подготовительный код перед запуском алгоритма.
new Population<BaseDoubleSpecies<SchwefelSpecies>> ();
...
private void CreateBtn_Click (object sender, EventArgs e)
{
// Количество хромосом (n в функции Швефеля)
int count = (int)chromoCount.Value;
// Зададим интервалы изменения хромосом
Interval[] intervals = new Interval[count];
for (int i = 0; i < count; i++)
{
intervals[i] = new Interval (-500.0, 500.0);
}
// Зададим параметры алгоритма
m_Population.Reset ();
m_Population.MaxSize = (int)popSize.Value;
m_Population.MutationPossibility = int.Parse (mutation.Text) / 100.0;
m_Population.CrossPossibility = int.Parse (cross.Text) / 100.0;
// Установим свойства видов
SchwefelSpecies.Intervals = intervals;
// Добавим виды
for (int i = 0; i < m_Population.MaxSize; ++i)
{
m_Population.Add (new SchwefelSpecies ());
}
}
Пример запуска этого проекта видно на следующих рисунках
Проект ConsoleSchwefelTest
Данный проект предназначен для более наглядной демонстрации использования класса BaseDoubleSpecies. В этом примере также минимизируется функция Швефеля от 15 переменных. Этот проект состоит из двух файлов: SchwefelSpecies.cs с классом особи и Program.cs с основной логикой работы генетического алгоритма.
Класс особи SchwefelSpecies очень простой и состоит только из расчета целевой функции (метод CalcFinalFunc()) и текстового представления особи (метод ToString()).
using System.Collections.Generic;
using System.Text;
using Jenyay.Genetic;
namespace ConsoleShwefelTest
{
class SchwefelSpecies: BaseDoubleSpecies<SchwefelSpecies>
{
public SchwefelSpecies ():
base ()
{
}
public SchwefelSpecies (double[] chromosomes):
base (chromosomes)
{
}
protected override double CalcFinalFunc ()
{
double result = 0;
for (int i = 0; i < m_Chromosomes.Length; i++)
{
result -=
m_Chromosomes[i] * Math.Sin (Math.Sqrt (Math.Abs (m_Chromosomes[i]) ) );
}
return result;
}
public override string ToString ()
{
StringBuilder builder = new StringBuilder();
for (int i = 0; i < m_Chromosomes.Length; i++)
{
builder.AppendFormat ("x[{0:D3}] = {1}\r\n", i, m_Chromosomes[i]);
}
builder.AppendLine ();
builder.AppendFormat ("FinalFunc = {0}", m_FuncVal);
return builder.ToString ();
}
}
}
Основная логика программы находится в файле Program.cs с подробными комментариями:
using System.Collections.Generic;
using System.Text;
using Jenyay.Genetic;
namespace ConsoleShwefelTest
{
class Program
{
static void Main (string[] args)
{
////////////////////////////////////////////////////////////////
// Входные параметры
////////////////////////////////////////////////////////////////
// Количество хромосом (неизвестных в функции Швефеля)
int chromosomesCount = 15;
// Максимальное количество поколений (итераций)
int maxGeneration = 10000;
// Максимальное количество поколений (итераций) при постоянном значении целевой функции
// Если за это количество поколений не найдено более лучшее решение, алгоритм прерывается.
int maxEqualGeneration = 300;
// Если на очередной итерации целевая функция изменилась на значение,
// меньшее ил равное minDelta, то считаем, что целевая функция не изменилась.
double minDelta = 1e-8;
// Максимальный размер популяции
int populationMaxSize = 100;
// Вероятность мутации
double mutationPossibility = 0.1;
// Вероятность скрещивания
double crossPossibility = 0.9;
// Интервал изменений хромосом
double chromoMinVal = -500;
double chromoMaxVal = 500;
////////////////////////////////////////////////////////////////
// Инициализацйия статических свойств класса SchwefelSpecies
InitializeSpecies (chromosomesCount, chromoMinVal, chromoMaxVal);
// Создание и инициализация популяции
Population<BaseDoubleSpecies<SchwefelSpecies>> population =
InitializePopulation (
populationMaxSize,
mutationPossibility,
crossPossibility);
// Основной цикл алгоритма
MainLoop (population, maxGeneration, maxEqualGeneration, minDelta);
Console.ReadKey ();
}
/// <summary>
/// Основной цикл алгоритма
/// </summary>
/// <param name="population">Созданная популяция</param>
/// <param name="maxGeneration">Максимальное количество поколений</param>
/// <param name="maxEqualGeneration">Максимальное количество поколений, когда значение целевой функции не изменилось</param>
/// <param name="minDelta">Минимальное изменение целевой функции, когда считаем, что найдено более лучшее решение</param>
private static void MainLoop (
Population<BaseDoubleSpecies<SchwefelSpecies>> population,
int maxGeneration,
int maxEqualGeneration,
double minDelta)
{
// Количество поколений, в течение которых значение целевой функции не изменилось
int equalGenerations = 0;
// Значение целевой функции на предыдущей итерации
double oldBestFinalFunc = population.BestFunc;
// Цикл, ограниченный максимальным номером поколения и
// количеством поколений, когда значение целевой функции не изменилось.
for (int generation = 0;
generation < maxGeneration && equalGenerations < maxEqualGeneration;
++generation)
{
// Вывод лучшей на данный момент особи в консоль
WriteBestSpeciesToConsole (population.BestSpecies, generation);
// Следующая итерация алгоритма. Получение следующего поколения особей.
population.NextGeneration ();
// Проверим, найдено ли лучшее решение
if (Math.Abs (population.BestFunc - oldBestFinalFunc) <= minDelta)
{
equalGenerations++;
}
else
{
oldBestFinalFunc = population.BestFunc;
}
}
}
/// <summary>
/// Инициализация класса особей. Изменяются статические свойства класса SchwefelSpecies
/// </summary>
/// <param name="chromosomesCount"></param>
/// <param name="chromoMinVal"></param>
/// <param name="chromoMaxVal"></param>
private static void InitializeSpecies (
int chromosomesCount,
double chromoMinVal,
double chromoMaxVal)
{
// Зададим интервалы изменения хромосом
Interval[] intervals = new Interval[chromosomesCount];
for (int i = 0; i < chromosomesCount; i++)
{
intervals[i] = new Interval (chromoMinVal, chromoMaxVal);
}
// Установим свойства видов
SchwefelSpecies.Intervals = intervals;
}
/// <summary>
/// Создание и инициализация популяции
/// </summary>
/// <param name="populationMaxSize">Максимальный размер популяции</param>
/// <param name="mutationPossibility">Вероятность мутации</param>
/// <param name="crossPossibility">Вероятность скрещивания</param>
/// <returns>Возвращает созданный экземпляр популяции.</returns>
private static Population<BaseDoubleSpecies<SchwefelSpecies>> InitializePopulation (
int populationMaxSize,
double mutationPossibility,
double crossPossibility)
{
Population<BaseDoubleSpecies<SchwefelSpecies>> population =
new Population<BaseDoubleSpecies<SchwefelSpecies>> ();
// Зададим параметры алгоритма
population.Reset ();
population.MaxSize = populationMaxSize;
population.MutationPossibility = mutationPossibility;
population.CrossPossibility = crossPossibility;
// Добавим виды
for (int i = 0; i < population.MaxSize; ++i)
{
population.Add (new SchwefelSpecies ());
}
return population;
}
/// <summary>
/// Вывод параметров особи в консоль
/// </summary>
/// <param name="species">Особь, параметры которой хотим вывести в консоль</param>
/// <param name="generation">Номер поколения, которой принадлежит особь</param>
private static void WriteBestSpeciesToConsole (
BaseDoubleSpecies<SchwefelSpecies> species,
int generation)
{
StringBuilder builder = new StringBuilder ();
builder.AppendLine (species.ToString ());
builder.AppendFormat ("Generation = {0}", generation);
builder.AppendLine ();
builder.AppendLine ();
Console.WriteLine (builder.ToString ());
}
}
}
Результат работы данного примера показан на следующем скриншоте:
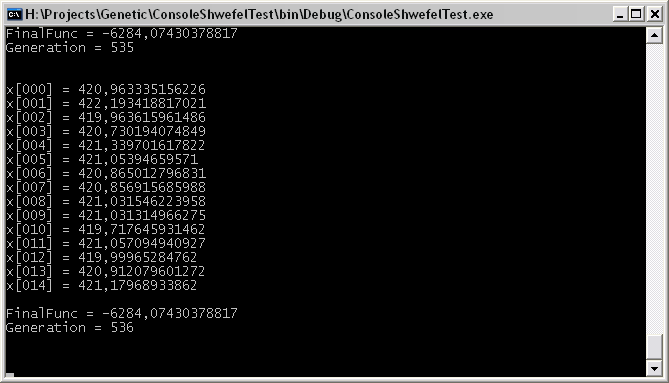
Вот, пожалуй, и все. Более подробно примеры использования смотрите в исходниках.
История версий
11.09.2011
- Добавил консольный пример ConsoleSchwefelTest.
24.06.2011
- Исправил ошибку в примере (спасибо Николаю за то, что ее нашел и не поленился о ней написать :)). Примеры сделал более наглядными.
2.2 (04.09.2009)
- Добавил класс Analytics.
- В примере SchwefelTest теперь выводится график сходимости алгоритма.
2.1 (24.08.2009)
- Добавил класс BaseDoubleSpecies для более удобной минимизации функций от дробных хромосом.
- Добавил проект, реализующий минимизацию функции Швефеля.
2.0
- Переписал библиотеку под .NET 2.0 с использованием обобщений (generics);
- В примере использования добавил возможность менять параметры алгоритма во время работы.
1.0
- Реализация под .NET 1.1.
Вы можете подписаться на новости сайта через RSS, Группу Вконтакте или Канал в Telegram.
